Surveillance de Salmonella en filière de fabrication de fromages au lait cru
| Groupe actif de 2017 à 2020 |
"Deux années de réflexions collectives au service de la surveillance des salmonelles"
Les premiers travaux de la plateforme SCA ont concerné la surveillance de Salmonella en filière de fabrication de fromages au lait cru.
Deux axes de travail ont été menés par un groupe rassemblant autorités, professionnels, instituts techniques, laboratoires et agences sanitaires : le premier a consisté à produire un document d'aide méthodologique pour la surveillance des Salmonella aux différentes étapes de la filière bovine de production de fromages au lait cru ; le second axe de travail a été la réalisation d’une étude de séquençage génomique sur 500 souches pour appréhender la circulation clonale de Salmonella Dublin dans une région donnée.
Un document d’aide méthodologique pour mieux surveiller Salmonella
Ce document a été conçu pour permettre aux professionnels d'améliorer la surveillance à chaque maillon de la chaîne alimentaire, pour renforcer les interactions et la communication entre ces maillons et ainsi obtenir une plus grande cohérence dans la mise en place des actions de surveillance.
Des annexes sur le plan d’échantillonnage et sur les méthodes analytiques, ainsi que des présentations de la plateforme SCA et du réseau Salmonella viennent compléter ce document.
Une étude couplant données écologiques et séquençage complet du génome
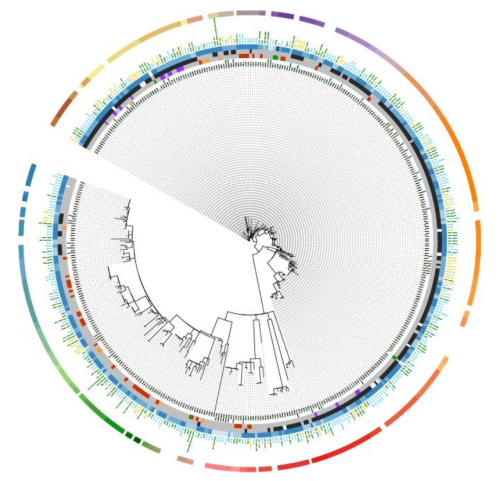
Co-financée par la DGAl et le Cniel, l’étude a été réalisée par une équipe constituée de personnels de l’Anses et d’Actalia. Des souches d’origine alimentaire et animale, ainsi que les données écologiques associées (métadonnées) ont été collectées avec la participation d’acteurs locaux. Une sélection de 500 souches a été séquencée par la méthode WGS (Whole Genome Sequencing). Les souches présentant une forte parenté génomique ont été regroupées en clusters. Associés aux métadonnées, ces groupes constitués permettent de rechercher des liens ou des flux entre les matrices tout au long de la chaîne alimentaire. Le rapport technique de cette étude présente la diversité génomique des souches de Salmonella Dublin isolées principalement en 2015 et 2016 en filière laitière franc-comtoise. Il souligne également les enseignements méthodologiques et perspectives dégagés par cette étude, qui a constitué le premier travail de ce type en configuration plateforme en France.
Groupes de Travail |
| Abstract |
| Ondes |
| SalmoSurv |
| Surveillance des STEC |

Groupes de Travail
Membres du Copil